Ein tiefer Blick ins Protein
Lichtmikroskope erlauben immer genauere Blicke in die Welt des Allerkleinsten. Nun haben Wissenschaftler der FAU und des Max-Planck-Instituts für die Physik des Lichts in Erlangen mit der sogenannten COLD-Methode erstmals Strukturen unterhalb eines Nanometers in einem Protein sichtbar gemacht. Diese Detailgenauigkeit erreicht COLD, weil es bei minus 270 Grad Celsius arbeitet und die Signale der leuchtenden Proteine bei diesen Temperaturen intensiver und damit genauer lokalisierbar werden. So lassen sich beispielsweise in vitro Strukturänderungen von Proteinen sichtbar machen, die mit Krankheiten assoziiert sind.
Bis Ende des 20. Jahrhunderts unterlagen Lichtmikroskope ausnahmslos einer Einschränkung: Die Wellennatur des Lichts begrenzte ihre Auflösung. Bereits 1873 hatte der Physiker Ernst Abbe dafür eine Gesetzmäßigkeit formuliert, die etwas vereinfacht lautet: Strukturen kleiner als die halbe Wellenlänge des sichtbaren Lichts – also rund 200 Nanometer – kann ein optisches Mikroskop nicht darstellen. „Das entspricht etwa einer Länge von mehreren tausend aneinandergereihten Atomen“, erklärt Vahid Sandoghdar, Inhaber des Lehrstuhls für Experimentalphysik an der FAU und gleichzeitig Direktor am Max-Planck-Institut für die Physik des Lichts.
Die Grenze galt in Expertenkreisen lange als unumstößlich – bis Forscher innerhalb der letzten zwanzig Jahre sogenannte ultrahochauflösende Verfahren der Fluoreszenzmikroskopie wie STED und PALM/STORM entwickelten. Dadurch verbesserten sie die optische Auflösung enorm, nämlich auf etwa 20 bis 30 Nanometer. Im Jahr 2014 bekamen die beteiligten Wissenschaftler, darunter Stefan Hell vom Max-Planck-Institut für biophysikalische Chemie in Göttingen, dafür den Nobelpreis für Chemie. Erst kürzlich steigerte ein Team um Hell mit der neu entwickelten MINFLUX-Mikroskopie die Auflösungsgrenze noch einmal um das 20-fache: Die Wissenschaftler machten benachbarte Moleküle sichtbar, die nur wenige Nanometer voneinander getrennt liegen.
COLD erlaubt den Blick ins Protein
Vahid Sandoghdar und seine Kollegen arbeiten seit einigen Jahren mit einer alternativen Methode ebenfalls daran, die Leistung der hochauflösenden Fluoreszenzmikroskopie zu steigern – mit Erfolg, wie der Erlanger Physiker jetzt berichtet: „Unsere Herangehensweise erlaubt es uns, Strukturen darzustellen, die weniger als fünf Ångström, also einen halben Nanometer, voneinander getrennt sind. Wir können daher gewissermaßen in ein Protein hineinsehen.“ Das zugrundeliegende Konzept stellten Sandoghdar und seine Kollegen erstmals im Jahr 2013 vor und tauften es COLD – eine Abkürzung basierend auf der englischen Bezeichnung Cryogenic Optical Localization in Three Dimensions. Der Name COLD (zu deutsch: kalt) ist Programm: „Das Entscheidende von COLD ist die niedrige Temperatur, bei der die Messungen stattfinden, rund minus 270 Grad Celsius“, so der Physiker.
Das grundlegende Prinzip ist unterdessen bei allen fluoreszenzmikroskopischen Methoden gleich: Man markiert die Probe zunächst chemisch mit fluoreszierenden Molekülen, Fluorophore genannt. So lassen sich etwa bestimmte Bereiche oder Komponenten von Zellen sichtbar machen. Die herkömmlichen Techniken werden dabei jedoch durch das Abbe-Limit beschränkt: Sobald mehrere nahe beieinander liegende Fluorophore gleichzeitig Licht emittieren, überlappen sich ihre Signale; man kann sie nicht als voneinander getrennt erkennen. Die hochauflösenden Methoden sind indes in der Lage, die Farbstoffmoleküle einzeln ein- und auch wieder auszuschalten. Dadurch lassen sich deren Positionen ziemlich genau bestimmen. Gleichwohl leidet die Präzision unter dem statistischen Rauschen; je kürzer die Fluorophore Licht aussenden, desto größer ist die Ungenauigkeit. Und üblicherweise ist das Leuchten nur von kurzer Dauer. Denn lichtinduzierte chemische Prozesse zerstören diese Fähigkeit.
Bei tiefen Temperaturen lassen sich Farbstoffe genauer lokalisieren
Hier setzt COLD an: Die extrem niedrigen Temperaturen verlangsamen die photochemischen Prozesse massiv, weshalb die Farbstoffe über einen deutlich längeren Zeitraum leuchten. „Dadurch verringert sich das Rauschen auf den Bildern und die Lokalisierungsgenauigkeit steigt“, erläutert Siegfried Weisenburger, der solche Experimente im Rahmen seiner Doktorarbeit in der Abteilung von Sandoghdar durchgeführt hat.
In Zusammenarbeit mit Christian Griesinger und seiner Abteilung am Max-Planck-Institut für biophysikalische Chemie in Göttingen konnten die Erlanger Wissenschaftler mit ihrer Technik zwei Fluorophore auf einem Protein auflösen, die nur einen Nanometer voneinander getrennt waren. Auf dieser Größenskala machen bereits geringste Vibrationen und Bewegungen der Probe das Experiment zunichte. „Um atomare Abstände messen zu können, mussten wir ein spezielles kryogenes Mikroskop entwickeln, das eine besonders hohe mechanische Stabilität aufweist. Für die Experimente werden die Proteine dann in einem dünnen Polymerfilm verteilt und auf einen Glasobjektträger aufgebracht“, berichtet Sandoghdar.
Wie sind Proteine in einer Membran ausgerichtet?
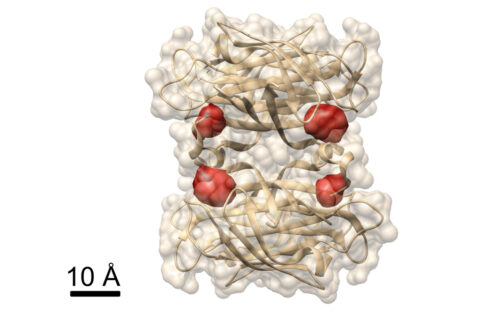
In einem weiteren Experiment demonstrierten die Forscher, dass man mit COLD die räumliche Struktur von Proteinen ermitteln kann: Sie markierten ein Protein namens Streptavidin an vier bestimmten Stellen mit Farbstoffen und nahmen mehrere hundert Bilder auf. Daraus rekonstruierten sie anschließend mittels Tomografie-Algorithmen, in welcher Struktur das Protein vorlag. Dabei erreichten sie letztlich eine dreidimensionale Auflösung von etwa einem halben Nanometer. „Dieses Auflösungsvermögen ermöglicht strukturbiologische Untersuchungen an der Grenze zu anderen strukturbiologischen Methoden.“ Dies gilt vor allem für das Spezialgebiet von Christian Griesinger: die NMR-Spektroskopie, die nur Distanzen unterhalb von fünf Ångström beobachten kann. „COLD schlägt damit erfolgreich eine Brücke von der Fluoreszenzmikroskopie zur Strukturbiologie“, so der Göttinger Max-Planck-Forscher.
Die Ergebnisse sind für eine Reihe von Wissenschaftlern sehr interessant, denn: „In Zellen ändern Proteine ihre Struktur, um ihre Aufgabe auszuführen oder ihre Funktion zu ändern. Ferner sind gewisse Strukturänderungen häufig mit bestimmten Krankheiten assoziiert“, wie Sandoghdar betont. Daneben kann COLD die Ausrichtung von Proteinen bestimmen. „Die Orientierung ist besonders dann interessant, wenn man Proteine in einer Membran untersucht. Das ist bereits jetzt Gegenstand unserer Experimente“, erklärt Daniel Böning, Mitglied des Erlanger Forscherteams.
Sandoghdar vermutet, dass sich die Auflösung durch weitere Optimierungen nochmal um das Zehnfache steigern lässt. Damit würde das Verfahren bis auf die atomare Ebene vordringen. Allerdings wird man dann an die intrinsische Grenze der Fluoreszenzmikroskopie stoßen, die aus der Größe der eingesetzten Farbstoffmoleküle resultiert. „COLD ist ideal, um Proteine oder kleinere Aggregate von Biomolekülen sichtbar zu machen. Gleichwohl kann die Technik wegen der unwirtlichen Temperaturen keine Vorgänge in lebenden Zellen untersuchen“, resümiert Sandoghdar und ergänzt: „Derzeit ist keine Mikroskopiemethode in der Lage, wirklich alles zu ‚sehen’. Die verschiedenen Strategien müssen daher – wie bislang und zumindest bis auf weiteres – stets komplementär zusammenarbeiten.“
Kontakt:
Prof. Dr. Vahid Sandoghdar
Tel.: 09131/7133-300
vahid.sandoghdar@mpl.mpg.de